使用Jmol制作分子轨迹动画以及旋转动画
Created At : 2019-08-19 00:00
Views 👀 :
原作者Yafan Zhao,本文转自zevan的博客
Jmol是一个强大的软件。使用Jmol可以很轻松的制作一些分子动画, 在Jmol官方wiki上有详细的介绍。
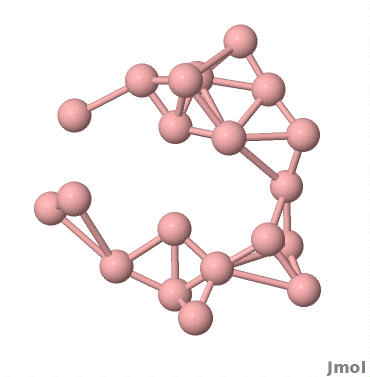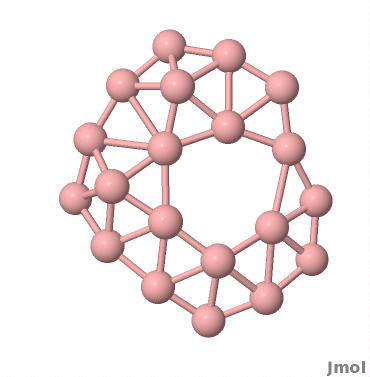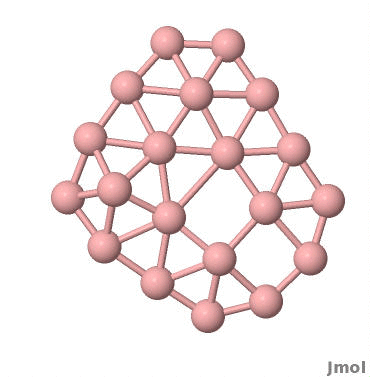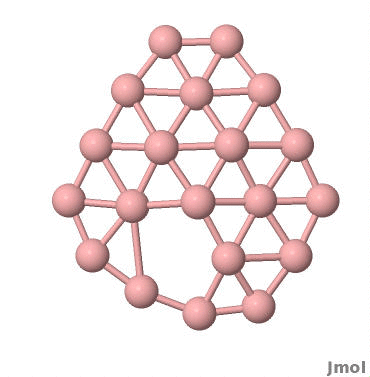
将几何优化或者分子动力学计算的轨迹展示为动画 首先,编写jmol脚本如下:
1 2 3 4 5 6 7 frame 1 num_frames = getProperty("modelInfo.modelCount") for (var i = 1; i <= num_frames; i = i+10) frame @i var filename = "movie"+("00000"+i)[-4][0]+".jpg" write IMAGE JPG @filename end for
将这段脚本保存为makemovie.jmol文件。使用Jmol打开轨迹文件,设置好颜色、背景、键的粗细、原子的大小等琐碎的细节。在Jmol的界面上点击文件->控制台,在出现的类似命令行终端下输入script makemovle.jmol,Jmol会输出一系列的JPG格式的图片。
1 convert -delay 10 movie00*.jpg B20-movie.gif
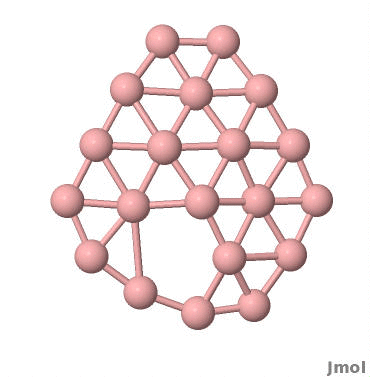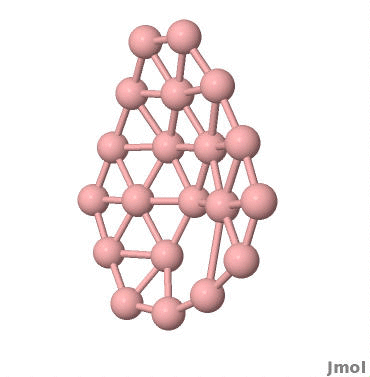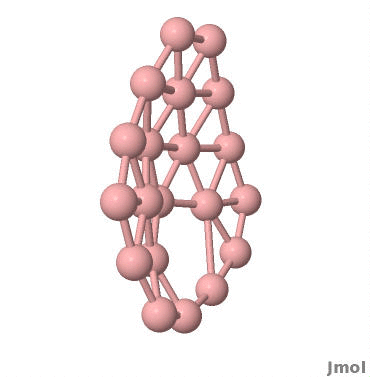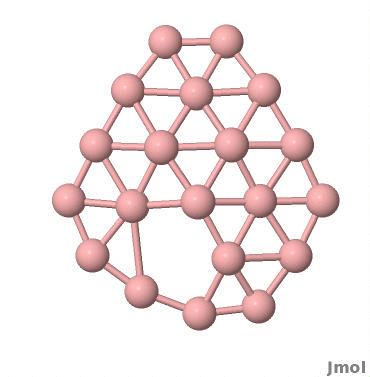
效果如图所示:
360度展示单个分子 类似的,编写jmol脚本如下:
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 # goal: a 360 degree spin ~degstep = 10 # or 2, degrees per step ~degrees = 0 # total degrees rotated while (~degrees < 360) { # SPIN rotate y @~degstep ~degrees += ~degstep # DISPLAY/WRITE FRAME refresh ~whereami = "degrees = " + ~degrees print ~whereami ~fileprefix = "Pic/" # UNCOMMENT TO GENERATE EXTRA FRAMES REQUIRED FOR WMV. COMMENT OUT FOR MP4. ~degreesplus = 1000 + ~degrees ~towrite = ~fileprefix + "B20_" + ~degreesplus + ".jpg" # WRITE THE IMAGE write image jpg @~towrite }
保存以上脚本为rotate.jmol文件。使用Jmol打开分子坐标文件,设置好颜色、背景、键的粗细、原子的大小等琐碎的细节。在当前文件夹下建立一个Pic文件夹。在控制台下输入 script rotate.jmol,Jmol会输出一系列的JPG格式的图片。
1 convert -delay 25 B20_*.jpg B20_anim.gif
效果如下:
参考文献:http://wiki.jmol.org/index.php/Creating_Movies
转载请注明来源,欢迎对文章中的引用来源进行考证,欢迎指出任何有错误或不够清晰的表达。